2010年ごろからITの世界では"ビッグ・データ時代の到来"などというキーワードを見かけるようになりました.テラバイト,ペタバイト,エクサバイト級の大規模データを高速に処理するためのソリューションが提案されています.技術的な背景としては, SSD(Solid State Drive)のような半導体ストレージの普及や64bit対応OSと大容量メモリを活用したインメモリデータベースなど,半導体技術の進歩があります.一方,大規模データを効率よく処理するための分散処理技術の進歩もあります.本稿で紹介するHadoopは,現時点で最も注目される分散処理技術の一つです.
筆者らは,蓄積された医療情報を活用するための基盤技術としてHadoopを活用することを検討しています.実際に試作システムの開発も開始しています.本稿では,生体モニタデータの分析を対象にしたHadoopの適用事例を紹介します.
Hadoopは分散処理フレームワーク(ソフトウェアの実行基盤)の一つです.Hadoopはインターネット検索サービス大手のGoogleが開発した技術をもとに実装された,オープンソースソフトウェアです. Yahoo!やFacebookなどのサービスプロバイダをはじめ,多くのシステムベンダーがHadoopの活用を進めています.
Hadoopは分散ファイルシステム(HDFS:Hadoop Distributed File System)と分散処理エンジン(MapReduce)から構成されています.従来から様々な分散処理技術が存在していましたが,Hadoopが注目されるようになった理由の一つにデータローカリティ機能があります.
図1に示すように,従来型の分散処理技術では,計算処理のためのコンピュータ(計算ノード)がディスクを共有していることがありました.計算処理が中心でディスクアクセスが少ない場合はこの方式でも十分機能します.しかし,大規模データの分析処理を行おうとすると共有ディスクへアクセスが増えるためボトルネックとなり,システム全体の処理性能が向上しません.
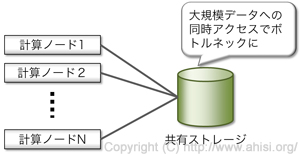
図1 従来型の分散処理
Hadoopでは図2に示すように,分散ファイルシステムと分散処理エンジンが同居する形になっています.ファイルは複数の計算ノードに分散して配置され,計算処理は対象のファイルが存在する計算ノード上で実行するように最適化されます.こうすることで,大規模データを効率よく処理することができるようになります.
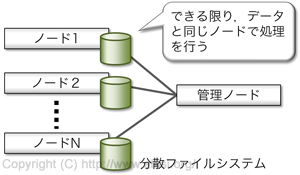
図2 Hadoopの分散処理
このほか,Amazon Elastic MapReduceのようなオンデマンドに利用できるクラウドサービスが存在する点もHadoopの特徴です.必要な時に必要な規模のHadoop環境を利用できます.利用した分だけ費用を支払えばよいため,研究機関や大規模病院に限られていたような高価なシステムを安価に利用できるようになります.
病院情報システムの電子化によって,院内で発生する様々な情報をデータとして蓄積できるようになってきました.次なるステップは蓄積されたデータの活用ですが,データからの知見の創出には複雑な計算処理が必要になるだけでなく,膨大なデータを効率よく活用できる仕組みが必要になります.Hadoopの適用は医療情報を対象にしたデータ活用において有用であると考えられます.
一般に,分散処理は計算処理におけるオーバヘッド(計算以外に必要となる処理)が大きいため,小規模な分析では通常のプログラムよりも計算時間を要することがあります.Hadoopの場合は分散ファイルシステムと分散処理エンジンが密接にかかわっているため,計算対象となるデータの粒度も処理時間に大きくかかわってくる可能性があります.
医療情報において,例えば検体検査の結果や生体モニタで計測された心拍数などは,患者ID,計測日時,計測値,計測単位といった数バイト程度のデータで構成されています.一方,放射線,MRI,超音波診断装置などからの画像は,数Mから数百Mバイト単位のデータとなります.このように,医療情報は大小さまざまな粒度のデータが存在しているという特徴があります.医療情報の効率的なデータ活用にHadoopを適用するには,どのような粒度でデータを構成し,どのように処理するかについての検討が重要となります.
筆者らは手始めとして,手術中の生体モニタデータを対象にした類似度分析にHadoopの導入を試みました.類似度の算出方法は様々ですが,本例では1分ごとの変化量を算出したのちに,これらのユーグリッド距離を類似度としました.
類似度の算出は症例の組合わせごとに行う必要があり,今回対象とした症例数(約6000件)では約1800万通りになります.すべての組合わせをあらかじめファイルとして保存しておこうとすると,総ファイルサイズは約14GBに相当します.
図3は,このような類似度計算をAmazon Elastic MapReduceを用いて実行したときの,CPU性能別の処理時間をまとめたものです. Amazon Elastic MapReduce はHadoopをオンデマンドに利用できるクラウドサービスです. 152ECU(2ECU注1×4仮想コアのマシン19台)の場合,142秒で処理を完了することができました.
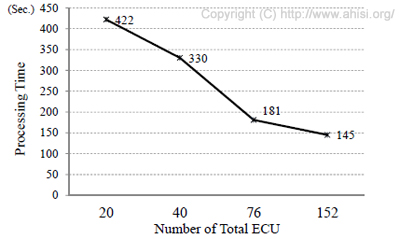
図3 Amazon Elastic MapReduceを用いた
Hadoopによる処理時間の例
比較のため,Hadoopを使わないプログラムを実装して,Amazon EC2上で同じ計算処理を行いました.2.5ECU注1×8仮想コアのマシン上で実行した結果を図4に示します.本プログラム自体も並列処理技術を実装して効率的な計算処理が可能になっていますが,並列処理数を4から8に増やしてもその効果が頭打ち傾向にあることがわかります.その原因として,ディスクアクセスに関わる時間があります.図中の点線で示したのは,計算処理中に占めるディスクアクセス時間の割合です.並列処理数が8の場合,全体の処理時間(402.9秒)のうち97.8%がディスクアクセス時間でした.すなわち,並列処理数を増やしてもディスクアクセスがボトルネックなっているため,計算処理全体の高速化が実現できないことを示しています.
したがって,仮に並列処理数を16,32・・・と増やしていってもHadoopで得られたような処理時間の短縮効果は見込めません.以上の結果は,Hadoopのデータローカリティ機能の有用性を示すものといえるでしょう.
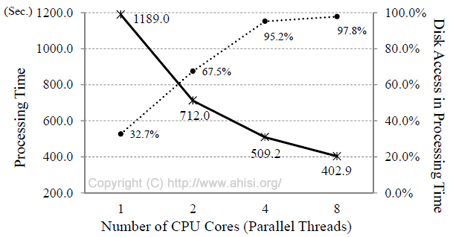
図4 非Hadoopプログラムにおける
処理時間とディスクアクセス
本稿ではHadoopの概要について説明いたしました.また, Hadoopのデータローカリティ機能の有用性を筆者らの実例をもとに説明いたしました.
言い換えれば,分析対象のデータが適度に分散されていることが,Hadoopによる効率的な分析のために重要であることになります. データが分散されていなければ,結果的にディスクアクセスが一定の場所に集中してボトルネックとなってしまいます.
前述のとおり,医療情報は大小さまざまな粒度のデータが雑多に混在しています.医療情報の利活用のためにHadoopを適用するには,このような雑多なデータをどのような単位でまとめ,どのように処理するかという議論がますます重要になります.筆者らは今後も医療情報に対するHadoopの活用について検討・実証をすすめ,本機構のホームページや学会発表などを通して成果を公開してまいります.
注1:CPU性能の単位であるECUについては,「ECU あたりのプロセッサー容量は、1.0-1.2 GHz の 2007 Opteron または 2007 Intel Xeon プロセッサーに相当します。」とAmazon AWSの各ページに明記されています.
- 水谷晃三,澤智博,医療情報分野でHadoopをどう使う?最新事情と効果的な利活用のためのアプローチ, Hadoop Conference Japan 2013 Winter.
・カンファレンスサイトはこちら.
・講演資料はこちら. - 水谷晃三,澤智博,テンプレートデータを用いた生体モニタデータの類似性分析に関する研究,第32回医療情報学連合大会,2-C-4-4,pp.658-659,2012.
- 水谷晃三,澤智博,Hadoopを活用した生体モニタデータの類似性分析システム,第16回医療情報学春期学術大会抄録集,PB-2-2-5,pp.184-185,2012.
- 水谷晃三,澤智博,分散処理フレームワーク"Hadoop"を用いた生体情報モニタデータ活用のための一方式,第31回医療情報学連合大会論文集,2-E-3-2,pp.541-542,2011.(若手奨励賞受賞)
(文責:水谷,2012-01-04掲載,2013-01-24一部更新)





